Evénement : Le forum France Culture, l’année vue par…les sciences
Par Pierre Chirsen
Par David Carriou - Le 6 mars 2013
La microscopie dite électronique permet d’obtenir des images d’échantillons pour une résolution inférieure au critère de Rayleigh (énonçant l’impossibilité d’observer, à l’aide de microscopes optiques, des entités dont la taille est inférieure à λ/2). Ces images sont des reconstructions informatiques des échantillons observés. Les plus connues sont le SEM (MEB : Microscope Electronique à Balayage) ou encore le TEM (MET : Microscopie Electronique en Transmission).
La Microscopie à Force Atomique (AFM) est également basée sur la reconstruction informatique des substrats analysés, cependant a contrario du MEB et du MET, il n’y a pas de préparations préalables à apporter et les échantillons n’ont pas besoin d’être mis sous vide secondaire voir tertiaire.
Ce type de microscope à sonde locale permet de visualiser la topographie de la surface d’un échantillon. Développé en 1985, par Binnig et al, il est essentiellement basé sur l’analyse d’un objet point par point au moyen d’un balayage via un microlevier ; balayage assuré par des céramiques piézoélectriques permettant des déplacements latéraux et verticaux de l’Angströms à plusieurs centaines de micromètres. Ce mode d’observation permet alors, de réaliser la cartographie locale des grandeurs physiques caractéristiques de l’objet sondé (force, courant, adhérence, module de Young,…), mais également de travailler dans des environnements particuliers tels que les milieux sous vide, liquides ou ambiants.
Un microscope à force atomique communément utilisé (Figure 1) est un AFM multimode Nanoscope VII (Bruker – Veeco Instruments, Santa Barbara, CA) associé à un dispositif de modulation de force.
Le schéma ci-dessous montre les principaux éléments d’un AFM, avec le dispositif de détection optique composé d’une diode laser et d’un photodétecteur, l’assemblage levier-pointe AFM, le tube piézo-électrique et l’électronique de rétrocontrôle.
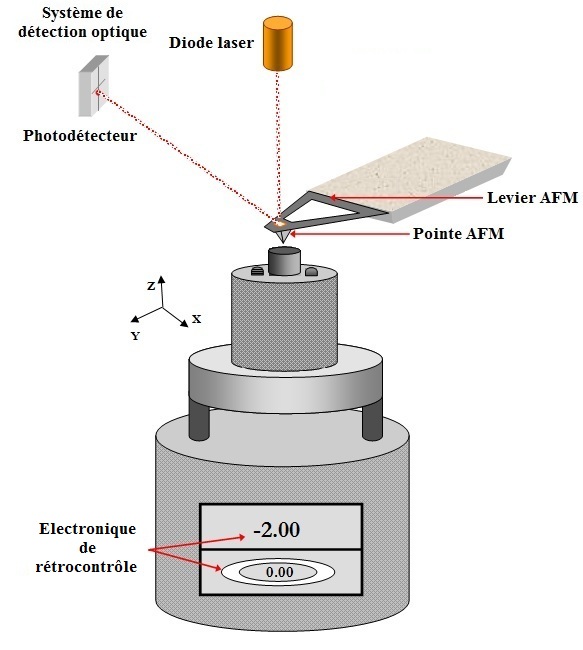
Figure 1 – Représentation des différents éléments constituant l’AFM.
Le fonctionnement du microscope à force atomique est basé sur la détection des forces qui s’exercent entre une pointe AFM intégrée à un levier et la surface d’un échantillon. L’AFM détecte les variations en déflexion du levier AFM à l’aide d’un dispositif optique composé d’une diode laser et d’un photodétecteur. Le débattement du levier est mesuré en focalisant un faisceau laser sur le levier, qui se réfléchit sur les quatre cadrans du photodétecteur. La séparation en quatre cadrans permet de discerner les angles de fléchissement normaux (représentant les forces normales à la surface de l’échantillon) et latéraux (traduisant les forces latérales).
L’AFM permet de mesurer des forces de l’ordre du piconewton (10-12 N). La force d’interaction entre les atomes de la pointe et ceux de la surface est maintenue constante au cours du balayage via une boucle de rétrocontrôle. La boucle de rétrocontrôle asservit la position pointe-échantillon de manière à maintenir constante cette interaction à la valeur de la force de consigne. L’enregistrement des mouvements (selon les directions X, Y et Z) du tube piézo-électrique donne alors une information topographique 3D de la surface. Les images obtenues en 512×512 (voire 1024×1024) points d’acquisition comportent une échelle de couleur. Les couleurs sombres représentent les parties basses de l’échantillon, alors que les couleurs claires traduisent les parties élevées de l’échantillon. L’acquisition d’une image se fait ligne par ligne à une fréquence de l’ordre du Hertz.
Il existe différents modes de fonctionnement pour l’AFM classique :
Mode AFM Contact:
La particularité du mode AFM Contact est de maintenir la pointe en contact avec l’échantillon sans aucune oscillation du levier. Ce mode permet d’obtenir l’image représentative de toute surface, de l’échelle micrométrique à l’échelle atomique. Il permet de mettre en évidence la friction créée par les phénomènes d’adhérence, d’élasticité locale ou par les différences de constitution chimique des échantillons observés en surface.
Mode Oscillant Contact:
Pour certains types d’échantillons, le mode AFM Contact peut être contraignant. En effet, les phénomènes de friction engendrés par le contact entre la pointe et l’échantillon peuvent endommager la surface étudiée, en particulier pour les échantillons biologiques.
Il a ainsi été développé un autre mode permettant de s’affranchir des forces de capillarité et de friction : le mode AFM oscillant Contact-Tapping. Le mode Tapping donne accès aux propriétés nanomécaniques des surfaces comme les variations des propriétés élastiques des matériaux, aussi bien à l’air qu’en milieu liquide.
Le système pointe-levier AFM possède un rôle majeur puisqu’il est à l’origine des mesures de forces. Les forces sont corrélées à la déflexion du levier.
Les principaux leviers AFM sont de forme rectangulaire (levier généralement en silicium monocristallin SiO2) ou de forme triangulaire (levier en nitrure de silicium Si3N4) (Figure 2). Un dépôt métallique Cr-Au est eff–ectué de manière à réfléchir au mieux le faisceau laser. Une pyramide, dont la hauteur se situe entre 2,5 et 8 µm et d’angle d’ouverture compris entre 15 et 25° suivant les faces, est réalisée à l’extrémité des leviers AFM. Selon le modèle de pointe AFM, l’extrémité peut être assimilée à une sphère de rayon variant entre 5 et 20 nm ou, dans le cas favorable, à un atome situé à l’extrémité d’une aspérité de la pointe.
Figure 2 – Images d’un levier triangulaire et d’un levier rectangulaire.
Il est évident de comprendre que la Microscopie à Force Atomique présente un rôle majeur dans l’étude d’échantillons biologiques, comme le présente ce cour historique.
La littérature met en évidence que l’observation des processus biologiques a commencé peu après l’invention de l’AFM en 1986 [1]. La première observation en milieu liquide, condition sine qua non à l’étude d’entités biologiques [2] fut réalisée en 1987 dans de l’huile de pétrole pour observer une surface de cristal de chlorure de sodium. Les avancées en microfabrication en 1988 au niveau de la fabrication de microlevier AFM et de l’utilisation d’une détection optique de la position du levier
(méthode OBD) [3] ont permis l’observation de cristaux d’acides aminés [4], de membranes lipidiques [5] ou encore d’immunoglobulines [6]. Un seul mode d’imagerie était pour l’heure disponible, le mode contact. Ce mode d’imagerie rendait difficile l’observation de processus dynamiques puisqu’il était trop intrusif pour les surfaces et les biomolécules faiblement liées. Le mode oscillant contact a été développé en 1993 [7]. Ce mode permet directement d’imager en liquide [8] et à haute résolution [9]. L’avantage majeur du mode de contact intermittent est qu’il conserve l’intégrité des surfaces même fragiles. Puis tout s’enchainera : observation d’ADN sur une surface de mica en 1994 [10], première image topographique haute résolution d’un transporteur transmembranaire, Ompf porin en 1995 [11], première observation du dépliement d’une seule protéine en 1997 [12] et description des changements conformationnels du dépliement d’une protéine en 1998 [13]. L’AFM ne s’utilise pas que pour des mesures topographiques mais également pour les mesures de forces d’interaction de tous types. Les interactions cellule – cellule via les couples ligand / récepteur des cellules ont été mesurées en 2000 [14]. En 2003, l’élasticité et l’adhérence de leucocytes furent mesurées [15] ainsi que la visco-élasticité des cellules épithéliales [16].
Un exemple de structure intéressante à étudier à l’AFM est la bacteriorhodopsin (Figure 3). La Bacteriorhodopsin est une protéine transmembranaire rencontrée chez les bactéries de la classe des Halobacteria, ayant un rôle de pompe à protons activée par la lumière et présente sous forme de « patch » dans leur membrane plasmique appelé membrane pourpre.
Des couches cytoplasmiques contenant bacteriorhodopsin ont été étudiées par AFM. Elles se présentent sous forme de trimères arrangés en treillis de 6.2 ± 0.2 nm de longueur [17]. Chaque sous- unité du trimère présente une extension de 0.8 ± 0.2 nm par rapport à la membrane lipidique.
Figure 3 – Illustration de la bacteriorhodopsin. (A) vue latérale du monomère, (B) vue cytoplasmique de la membrane pourpre et (C) image topographique.
Binnig, G., C.F. Quate, and C. Gerber, Atomic Force Microscope. Physical Review Letters, 1986. 56(9): p. 930.
Marti, et al., Atomic force microscopy of liquid-covered surfaces: atomic resolution images. Vol. 51. 1987, Melville,
NY, ETATS-UNIS: American Institute of Physics.
Meyer, et al., Novel optical approach to atomic force microscopy. Vol. 53. 1988, Melville, NY, ETATS-UNIS:
American Institute of Physics.
Gould, et al., Molecular resolution images of amino acid crystals with the atomic force microscope. Vol. 332. 1988,
London, ROYAUME-UNI: Nature Publishing Group.
Marti, et al., Scanning probe microscopy of biological samples and other surfaces. 1988, Oxford, ROYAUME-UNI:
Blackwell. 7.
Lin, et al., Direct observation of immunoglobulin adsorption dynamics using the atomic force microscope. Vol. 6.
1990, Washington, DC, ETATS-UNIS: American Chemical Society. 3.
Zhong, et al., Fractured polymer/silica fiber surface studied by tapping mode atomic force microscopy. Vol. 290.
1993, Kidlington, ROYAUME-UNI: Elsevier.
Putman, et al., Viscoelasticity of living cells allows high resolution imaging by tapping mode atomic force
microscopy. Vol. 67. 1994, Cambridge, MA, ETATS-UNIS: Cell Press. 5.
Moller, et al., Tapping-mode atomic force microscopy produces faithful high-resolution images of protein surfaces.
Vol. 77. 1999, Cambridge, MA, ETATS-UNIS: Cell Press. 9.
Bezanilla, et al., Motion and enzymatic degradation of DNA in the atomic force microscope. Vol. 67. 1994,
Cambridge, MA, ETATS-UNIS: Cell Press. 6.
Schabert, et al., Native Escherichia coli OmpF porin surfaces probed by atomic force microscopy. Vol. 268. 1995,
Washington, DC, ETATS-UNIS: American Association for the Advancement of Science.
Rief, M., et al., Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science, 1997.
276(5315): p. 1109-1112.
Marszalek, P.E., et al., Polysaccharide elasticity governed by chair-boat transitions of the glucopyranose ring.
Nature, 1998. 396(6712): p. 661-664.
Benoit, M., et al., Discrete interactions in cell adhesion measured by single-molecule force spectroscopy. Nat Cell
Biol, 2000. 2(6): p. 313-317.
Wojcikiewicz, E.P., et al., Contributions of molecular binding events and cellular compliance to the modulation of
leukocyte adhesion. J Cell Sci, 2003: p. jcs.00465.
Alcaraz, J., et al., Microrheology of Human Lung Epithelial Cells Measured by Atomic Force Microscopy. Biophysical
journal, 2003. 84(3): p. 2071-2079.
Müller, D.J., et al., Imaging purple membranes in aqueous solutions at sub-nanometer resolution by atomic force
microscopy. Biophysical journal, 1995. 68(5): p. 1681-1686.
Toi aussi participe au blog !
ParticiperPar Pierre Chirsen
Par Karine Da Silva Lages
Par Clément Dufrenne
Par Mélissa COURT